คริสเปอร์
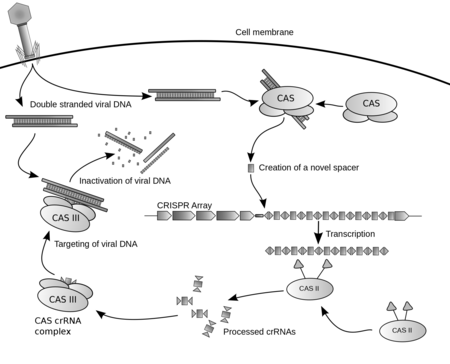
คริสเปอร์ (อังกฤษ: CRISPR) เป็นวงศ์ของลำดับดีเอ็นเอในแบคทีเรีย ซึ่งในลำดับมีส่วนของดีเอ็นเอ (DNA snippets) จากไวรัสที่เคยโจมตีแบคทีเรียนั้น แบคทีเรียใช้ส่วนเหล่านี้ในการตรวจหาและทำลายดีเอ็นเอจากไวรัสที่คล้ายกันในการโจมตีครั้งถัดไป ลำดับเหล่านี้ทำหน้าที่สำคัญในระบบภูมิคุ้มกันของแบคทีเรีย และเป็นพื้นฐานของเทคโนโลยีที่เรียกว่า คริสเปอร์/แคสไนน์ (อังกฤษ: CRISPR/Cas9) ที่สามารถใช้เปลี่ยนยีนภายในสิ่งมีชีวิต
ระบบคริสเปอร์/แคสเป็นระบบภูมิคุ้มกันโพรแคริโอตที่ช่วยในการต่อต้านส่วนประกอบทางพันธุกรรมแปลกปลอม เช่นเดียวกับที่อยู่ในพลาสมิด (plasmid) และ เฟจ (phages) ซึ่งเป็นภูมิคุ้มกันที่เกิดขึ้นภายหลัง (acquired immunity) อาร์เอ็นที่มีลำดับสเปสเซอร์ (spacer sequence) ช่วยโปรตีนแคส (CRISPR-associated, Cas) ในการจำแนกและตัดดีเอ็นเอจากภายนอก ขณะที่โปรตีนแคสอื่นที่นำโดยอาร์เอ็นเอตัดอาร์เอ็นเอจากภายนอก คริสเปอร์ถูกพบในประมาณ 40% ของจีโนมแบคทีเรียและใน 90% ของอาร์เคียที่ถูกหาลำดับดีเอ็นเอแล้ว
CRISPR ย่อมาจาก Clustered Regularly Interspaced Short Palindromic Repeats ชื่อถูกตั้งก่อนจะรู้ที่มาและหน้าที่ของลำดับย่อยอินเตอร์สเปส (interspacing subsequence) ณ ตอนนั้น คริสเปอร์ถูกมองว่าเป็นส่วนหนึ่งของดีเอ็นเอโพรแคริโอตที่มีลำดับเบสสั้น ๆ ที่เรียงตัวซ้ำ ๆ ลำดับซ้ำพาลินโดรม (palindromic repeat) เป็นลำดับนิวคลีโอไทด์ที่เหมือนกันเมื่ออ่านจากทั้งสองด้าน การซ้ำแต่ละครั้งตามด้วยดีเอ็นเอสเปสเซอร์จากการพบกับดีเอ็นเอภายนอกครั้งก่อน (เช่น ไวรัส หรือ พลาสมิด) ต่อจากลำดับคริสเปอร์ก็มีกลุ่มเล็ก ๆ ของยีนแคส (CRISPR-associated system, cas)
ระบบคริสเปอร์/แคสแบบง่ายที่เรียกว่าคริสเปอร์/แคสไนน์ถูกปรับแต่งเพื่อแก้ไขจีโนม จีโนมสามารถถูกตัดในตำแหน่งที่ต้องการ โดยการส่งแคสไนน์นิวคลีเอส (Cas9 nuclease) ประกอบกับไกด์อาร์เอ็นเอ (guide RNA, gRNA) เข้าไปในเซลล์ ทำให้สามารถกำจัดยีนที่มีอยู่หรือเพิ่มยีนใหม่เข้าไปได้ กลุ่มรวม Cas9-gRNA ทำหน้าที่เหมือน กลุ่มรวม CAS III crRNA บนแผนภาพด้านข้าง
เทคนิคการแก้ไขจีโนมคริสเปอร์/แคสมีศักยภาพในการนำไปประยุกต์ใช้ในหลายด้าน รวมถึงทางการแพทย์ และทางการปรุงแต่งเมล็ดพืชทางเกษตร การใช้กลุ่มรวม CRISPR/Cas9-gRNA เพื่อแก้ไขจีโจม ถูกเลือกโดยสมาคมเพื่อความก้าวหน้าทางวิทยาศาสตร์อเมริกัน (AAA) ให้เป็นการค้นพบครั้งใหญ่ใน พ.ศ. 2558 ข้อกังวลด้านชีวจริยธรรมถูกกล่าวถึงเกี่ยวกับความเป็นไปได้ในการใช้เทคนิคนี้เพื่อแก้ไขเซลล์สืบพันธุ์
ประวัติ
การค้นพบของกลุ่มดีเอ็นเอลำดับซ้ำเกิดขึ้นโดยอิสระในสามส่วนของโลก หนึ่งในการค้นพบครั้งแรกเกิดขึ้นเมื่อ พ.ศ. 2530 ณ มหาวิทยาลัยโอซะกะในประเทศญี่ปุ่น นักวิจัย โยชิซุมิ อิชิโนะ (Yoshizumi Ishino) และคณะเผยแพร่ผลการทดลองเกี่ยวกับลำดับของยีนชื่อว่า "iap" และความเกี่ยวข้องกับ E. coli ความก้าวหน้าทางเทคโนโลยีในช่วงคริสต์ทศวรรษ 1990 ทำให้พวกเขาสามารถดำเนินงานวิจัยต่อและเพิ่มความเร็วของการหาลำดับด้วยเทคนิคที่เรียกว่าเมตาจีโนมิกส์ (metagenomics) พวกเขาสามารถเก็บตัวอย่างน้ำทะเลหรือดินและหาลำดับดีเอ็นเอในตัวอย่างนั้น
กลไก

ภูมิคุ้มกันคริสเปอร์-แคสเป็นกระบวนการทางธรรมชาติของแบคทีเรียและอาร์เคีย คริสเปอร์-แคสป้องกันการติดเชื้อเฟจ, คอนจูเกชัน (conjugation) และการแปลงพันธุ์ทางธรรมชาติ โดยการทำลายกรดนิวคลีอิกจากภายนอกที่เข้ามาในเซลล์
การได้มาซึ่งสเปสเซอร์
เมื่อจุลินทรีย์ถูกบุกรุกโดยไวรัส ขั้นแรกของการตอบสนองของภูมิคุ้มกันคือการจับดีเอ็นเอของไวรัสและใส่เข้าไปในโลคัสคริสเปอร์ในรูปแบบของสเปสเซอร์ การที่ Cas1 และ Cas2 อยู่ในระบบภูมิคุ้มกันคริสเปอร์-แคสทั้งสองแบบ ชี้ว่าพวกมันมีส่วนในการได้มาซึ่งสเปสเซอร์ งานวิจัยการกลายพันธุ์ยืนยันสมมติฐานนี้ และแสดงว่าการนำยีน cas1 หรือ cas2 ออกทำให้ไม่สามารถจัดหาสเปสเซอร์ได้ ในขณะที่ไม่ส่งผลกระทบต่อการตอบสนองของภูมิคุ้มกันคริสเปอร์
โปรตีน Cas1 หลายโปรตีนถูกศึกษาลักษณะเฉพาะและหาโครงสร้าง โปรตีน Cas1 มีลำดับกรดอะมิโนหลากหลาย อย่างไรก็ตาม โครงสร้างผลึกมีลักษณะคล้ายกัน และโปรตีน Cas1 ที่ถูกทำให้บริสุทธิ์ทั้งหมดเป็นนิวคลีเอส/อินทีเกรสที่ต้องพึ่งธาตุโลหะ และจับกับดีเอ็นเอโดยไม่พึ่งกับลำดับ โปรตีน Cas2 ที่ถูกเลือกได้รับการศึกษาลักษณะเฉพาะ และพบว่าพวกมันแสดงกิจกรรมเอนโดริโบนิวคลีเอสเฉพาะต่อ ssRNA (สายเดี่ยว) หรือ dsDNA (สายคู่)
ในระบบไอ-อี ของ E. coli Cas1 และ Cas 2 จับกันเป็นกลุ่มรวมโดยสองส่วน (dimer) ของ Cas2 จับเข้ากับสองส่วนของ Cas1 ในกลุ่มรวมนี้ Cas2 มีหน้าที่เป็นโครงสร้างแบบไม่ใช้เอนไซม์ และจับกับชิ้นสายคู่ของดีเอ็นเอจากภายนอก ขณะที่ Cas1 จับกับด้านข้างของดีเอ็เอส่วนสายเดี่ยวและช่วยเร่งในการรวมเข้ากับแถวลำดับคริสเปอร์ สเปสเซอร์ใหม่ถูกเพิ่มในส่วนเริ่มของคริสเปอร์ต่อจากลำดับนำ (leader sequence) ทำให้เป็นการบันทึกการติดเชื้อไวรัสตามลำดับเวลา
โมทีฟติดกับโปรโตสเปสเซอร์
การวิเคราะห์ทางชีวสารสนเทศศาสตร์บริเวณจีโนมเฟจที่ถูกตัดเป็นสเปสเซอร์ (เรียกว่าโปรโตสเปสเซอร์, protospacer) ชี้ว่าไม่ได้เป็นการเลือกอย่างสุ่ม ทว่าถูกพบติดกับลำดับดีเอ็นเอสั้น ๆ (3 – 5 คู่เบส) ที่เรียกว่า protospacer adjacent motifs หรือ แพม (PAM) การวิเคราะห์ของระบบคริสเปอร์-แคสแสดงว่าลำดับแพมมีความสำคัญสำหรับระบบแบบที่ 1 (type I) และแบบที่ 2 (type II) ทว่าไม่สำคัญในแบบที่ 3 (type III) ระหว่างการได้มาซึ่งสเปสเซอร์ ในระบบแบบที่ 1 และ 2 โปรโตสเปสเซอร์ถูกตัดบนตำแหน่งติดกับลำดับแพม โดยอีกด้านหนึ่งของสเปสเซอร์ถูกตัดโดย ruler mechanism ทำให้ขนาดของสเปสเซอร์ใกล้เคียงกันในแถวลำดับคริสเปอร์ การอนุรักษ์ลำดับแพมแตกต่างกันไปในคริสเปอร์-แคสแต่ละระบบและอาจเชื่อมโยงกับ Cas1 และลำดับนำ (leader sequence)
สเปสเซอร์ใหม่ถูกเพิ่มเข้าไปในแถวลำดับคริสเปอร์โดยมีทิศทางที่แน่นอน มักเลือกที่จะ แต่ไม่เพียงแต่ ติดกับลำดับนำ การวิเคราห์ของระบบไอ-อีจาก E. coli แสดงว่าลำดับซ้ำแรกติดกับลำดับนำถูกคัดลอก โดยมี สเปสเซอร์ใหม่ที่พึ่งได้มาแทรกระหว่างลำดับซ้ำแรกและสอง
ลำดับแพมน่าจะมีความสำคัญระหว่างการแทรกสเปสเซอร์ในระบบไอ-อี ลำดับนั้นมีส่วนประกอบของนิวคลีโอไทด์ท้ายที่ถูกคงไว้ติดกับนิวคลีโอไทด์แรกของโปรโตสเปสเซอร์ นิวครีโอไทด์นี้กลายเป็นเบสสุดท้ายของลำดับซ้ำโดยตรง (direct repeat) ชิ้นแรก สิ่งนี้ชี้ว่ากลไกการได้มาซึ่งสเปสเซอร์ทำให้เกิดส่วนยื่นสายเดี่ยวในตำแหน่งรองท้ายของลำดับซ้ำโดยตรงและลำดับแพมระหว่างการแทรกสเปสเซอร์ อย่างไรก็ตาม ไม่ใช่ทุกแบบของคริสเปอร์-แคสจะใช้กลไกนี้ ด้วยความที่แพมในสิ่งมีชีวิตอื่นไม่แสดงการคงไว้ของตำแหน่งสุดท้ายในระดับเท่ากัน เป็นไปได้ว่าระบบเหล่านี้ทำให้เกิดปลายทู่ที่ไม่มีส่วนยื่นออกมาตรงส่วนท้ายของลำดับซ้ำโดยตรงและโปรโตสเปสเซอร์ระหว่างการได้มาซี่งสเปสเซอร์
การประยุกต์
ภายใน พ.ศ. 2557 มีงานวิจัยกว่า 1,000 งานที่กล่าวถึงคริสเปอร์ถูกตีพิมพ์ เทคโนโลยีถูกใช้เพื่อหยุดยั้งการปฏิบัติงานของยีนในเซลล์ไลน์มนุษย์และในเซลล์, เพื่อศึกษา Candida albicans, เพื่อปรับแต่งยีสต์เพื่อผลิตเชื้อเพลิงชีวภาพ และเพื่อปรับแต่งพันธุกรรมของสายพันธุ์พืช คริสเปอร์ยังสามารถถูกใช้เพื่อเปลี่ยนยุงให้ไม่สามารถถ่ายทอดโรค เช่น มาลาเรีย
การประเมินคำอ้างใหม่สำหรับความสัมพันธ์ระหว่างยีนกับโรคบนฐานของคริสเปอร์นำไปสู้การค้นพบความผิดปกติที่อาจมีความสำคัญ
พันธุวิศวกรรม
การแก้ไขจีโนมโดยคริสเปอร์/แคสไนน์ใช้ระบบคริสเปอร์แบบที่ 2 โดยใช้ Cas9, crRNA, tracrRNA และอาจใช้ร่วมกับส่วนของต้นแบบการซ่อมแซมดีเอ็นเอเพื่อช่วยในการเชื่อมต่อชิ้นส่วนโครโมโซมที่ไม่ใช่คู่ของมันเอง (non-homologous end joining, NHEJ) หรือ homology directed repair (HDR) ในการแก้ไขจีโนม
ส่วนประกอบหลัก
| ส่วรประกอบ | หน้าที่ |
|---|---|
| crRNA | มีไกด์อาร์เอ็นเอซึ่งหาที่ตั้งของดีเอ็นเอของโฮสต์พร้อมกับตำแหน่ที่จับกับ tracrRNA (มักอยู่ในรูปห่วง, hairpin loop) เกิดเป็นกลุ่มรวมพร้อมปฏิบัติการ |
| tracrRNA | จับกับ crRNA และเกิดเป็นกลุ่มรวมพร้อมปฏิบัติการ |
| sgRNA | ย่อมาจาก Single guide RNAs เป็นการรวมกันของอาร์เอ็นเอประกอบด้วย tracrRNA และ crRNA อย่างน้อยหนึ่งชิ้น |
| Cas9 | โปรตีนที่รูปแบบพร้อมปฏิบัติการสามารถปรับแต่งดีเอ็นเอได้ มีหลายรูปแบบที่มีหน้าที่แตกต่างกัน (เช่น การตัดสายเดี่ยว การตัดสายคู่ การจับกับดีเอ็นเอ) เป็นผลจากหน้าที่ของ Cas9 ในการรู้จำตำแหน่งดีเอ็นเอ |
| ต้นแบบการซ่อมแซม (repair template) | ดีเอ็นเอที่นำกระบวนการซ่อมแซมเซลทำให้สามารถแทรกลำดับดีเอ็นเอเฉพาะได้ |
คริสเปอร์/แคสไนน์มักใช้พลาสมิดเพื่อบุกรุก (transfect) เซลล์เป้าหมาย ส่วนประกอบหลักของพลาสมิดแสดงอยู่ในรูปทางขวา เริ่มจากการออกแบบ crRNA สำหรับการประยุกต์ใช้แต่ละครั้งด้วยความที่สิ่งนี้เป็นลำดับที่ Cas9 ใช้เพื่อระบุและจับโดยตรงกับดีเอ็นเอของเซลล์ crDNA ต้องจับกับที่ซึ่งต้องการแก้ไขเท่านั้น ต้นแบบการซ่อมแซมถูกออกแบบสำหรับการใช้แต่ละครั้งด้วยความที่ต้องทับซ้อนกับด้านใดด้านหนึ่งที่ถูกตัดและต้องเป็นรหัสสำหรับการแทรกลำดับ
crRNAs และ tracrRNA หลายชิ้นสามารถบรรจุเข้าด้วยกันเพื่อสร้าง single-guide RNA (sgRNA) โดยสามารถนำ sgRNA นี้ไปต่อกับยีน Cas9 ในพลาสมิดเพื่อบุกรุกเข้าสู่เซลล์
ดูเพิ่ม
-
Doudna, Jennifer; Mali, Prashant (23 March 2016). CRISPR-Cas: A Laboratory Manual. New York: Cold Spring Harbor Laboratory Press. ISBN 978-1-62182-131-1.
{{cite book}}: ไม่รู้จักพารามิเตอร์|name-list-format=ถูกละเว้น แนะนำ (|name-list-style=) (help) - Mohanraju P, Makarova KS, Zetsche B, Zhang F, Koonin EV, van der Oost J (August 2016). "Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems". Science. 353 (6299): aad5147. doi:10.1126/science.aad5147. PMID 27493190.
- Sander JD, Joung JK (April 2014). "CRISPR-Cas systems for editing, regulating and targeting genomes". Nature Biotechnology. 32 (4): 347–55. doi:10.1038/nbt.2842. PMC 4022601. PMID 24584096.
- Slaymaker IM, Gao L, Zetsche B, Scott DA, Yan WX, Zhang F (January 2016). "Rationally engineered Cas9 nucleases with improved specificity". Science. 351 (6268): 84–8. doi:10.1126/science.aad5227. PMC 4714946. PMID 26628643.
- Terns RM, Terns MP (March 2014). "CRISPR-based technologies: prokaryotic defense weapons repurposed". Trends in Genetics. 30 (3): 111–8. doi:10.1016/j.tig.2014.01.003. PMC 3981743. PMID 24555991.
- Westra ER, Buckling A, Fineran PC (May 2014). "CRISPR-Cas systems: beyond adaptive immunity". Nature Reviews Microbiology. 12 (5): 317–26. doi:10.1038/nrmicro3241. PMID 24704746.
- Andersson AF, Banfield JF (May 2008). "Virus population dynamics and acquired virus resistance in natural microbial communities". Science. 320 (5879): 1047–50. Bibcode:2008Sci...320.1047A. doi:10.1126/science.1157358. PMID 18497291.
- Hale C, Kleppe K, Terns RM, Terns MP (December 2008). "Prokaryotic silencing (psi) RNAs in Pyrococcus furiosus". RNA. 14 (12): 2572–9. doi:10.1261/rna.1246808. PMC 2590957. PMID 18971321.
- van der Ploeg JR (June 2009). "Analysis of CRISPR in Streptococcus mutans suggests frequent occurrence of acquired immunity against infection by M102-like bacteriophages". Microbiology. 155 (Pt 6): 1966–76. doi:10.1099/mic.0.027508-0. PMID 19383692.
- van der Oost J, Brouns SJ (November 2009). "RNAi: prokaryotes get in on the act". Cell. 139 (5): 863–5. doi:10.1016/j.cell.2009.11.018. PMID 19945373.
- Karginov FV, Hannon GJ (January 2010). "The CRISPR system: small RNA-guided defense in bacteria and archaea". Molecular Cell. 37 (1): 7–19. doi:10.1016/j.molcel.2009.12.033. PMC 2819186. PMID 20129051.
- Pul U, Wurm R, Arslan Z, Geissen R, Hofmann N, Wagner R (March 2010). "Identification and characterization of E. coli CRISPR-cas promoters and their silencing by H-NS". Molecular Microbiology. 75 (6): 1495–512. doi:10.1111/j.1365-2958.2010.07073.x. PMID 20132443.
- Díez-Villaseñor C, Almendros C, García-Martínez J, Mojica FJ (May 2010). "Diversity of CRISPR loci in Escherichia coli". Microbiology. 156 (Pt 5): 1351–61. doi:10.1099/mic.0.036046-0. PMID 20133361.
- Deveau H, Garneau JE, Moineau S (2010). "CRISPR/Cas system and its role in phage-bacteria interactions". Annual Review of Microbiology. 64: 475–93. doi:10.1146/annurev.micro.112408.134123. PMID 20528693.
- Koonin EV, Makarova KS (December 2009). "CRISPR-Cas: an adaptive immunity system in prokaryotes". F1000 Biology Reports. 1: 95. doi:10.3410/B1-95. PMC 2884157. PMID 20556198.
- "The age of the red pen". The Economist. August 22, 2015. ISSN 0013-0613. สืบค้นเมื่อ 2015-08-25.